co2 <- read.csv("co2.csv", header=FALSE)
head(co2) V1 V2 V3
1 2024/5/17 17:41:15 633
2 2024/5/17 17:41:18 633
3 2024/5/17 17:41:27 637
4 2024/5/17 17:41:30 638
5 2024/5/17 17:41:33 639
6 2024/5/17 17:41:36 639CSVファイルはread.csv()で読み込むことができます。 この函数は、data.frameと呼ばれる、リストに似たオブジェクト返します。 行や列に名前を付けて参照することができます。
二酸化炭素の測定をして、1列目が日付、2列目が時刻、3列目が測定値というCSVファイルが得られたとします。
co2 <- read.csv("co2.csv", header=FALSE)
head(co2) V1 V2 V3
1 2024/5/17 17:41:15 633
2 2024/5/17 17:41:18 633
3 2024/5/17 17:41:27 637
4 2024/5/17 17:41:30 638
5 2024/5/17 17:41:33 639
6 2024/5/17 17:41:36 639列に名前を付けましょう。
names(co2) <- c("date", "time", "co2")名前を使ってco2$dateまたはco2[["date"]]とすると、列をベクトルとして取得できます。 co2$["date"]はdateを残したdata.frameを返します。
日付と時刻をPOSIXctに変換しておくと、時系列データを扱うときに便利です。 POSIXctはRの日時オブジェクトの一つで、基準時刻からの秒数で表します。 ctはcalendar timeを意味します。 POSIXltはlocal timeで要素を整数のベクトルで表します。
co2$datetime <- as.POSIXct(paste(co2$date, co2$time))
head(co2) date time co2 datetime
1 2024/5/17 17:41:15 633 2024-05-17 17:41:15
2 2024/5/17 17:41:18 633 2024-05-17 17:41:18
3 2024/5/17 17:41:27 637 2024-05-17 17:41:27
4 2024/5/17 17:41:30 638 2024-05-17 17:41:30
5 2024/5/17 17:41:33 639 2024-05-17 17:41:33
6 2024/5/17 17:41:36 639 2024-05-17 17:41:36不要な列はNULLを代入して削除します。
co2$date <- NULL
co2$time <- NULL時間変化をプロットしてみましょう。
plot(co2$datetime, co2$co2, type="l", xlab="Date", ylab="CO2 concentration")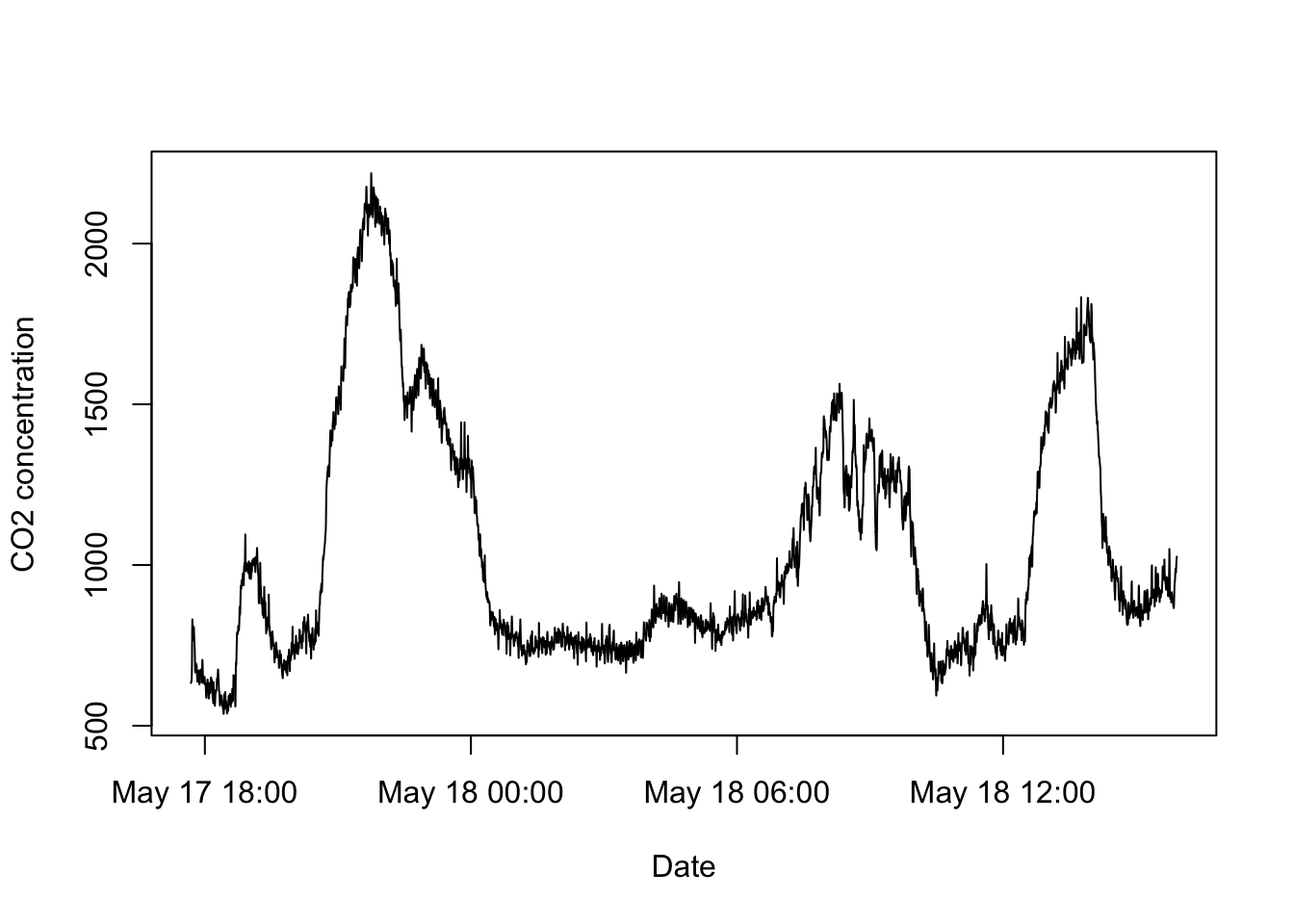
type="l"は線グラフを指定します。xlabとylabで軸のラベルを指定します。
頻度分布を調べるために、ヒストグラムを描いてみましょう。
hist(co2$co2, breaks=20, main="CO2 concentration histogram", xlab="CO2 concentration")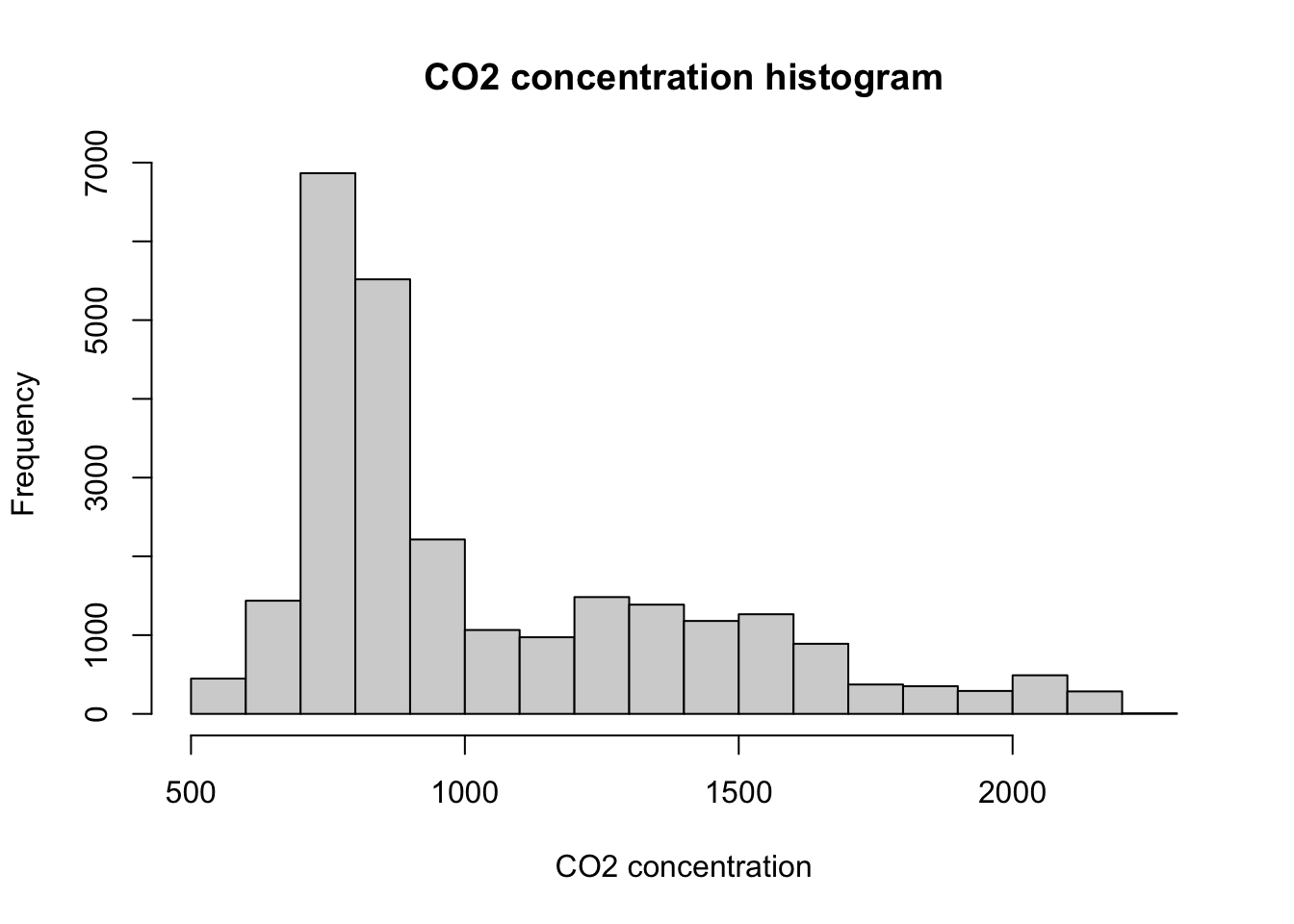
箱ひげ図を描いてみましょう。
bp <- boxplot(co2$co2, main="CO2 concentration boxplot", ylab="CO2 concentration")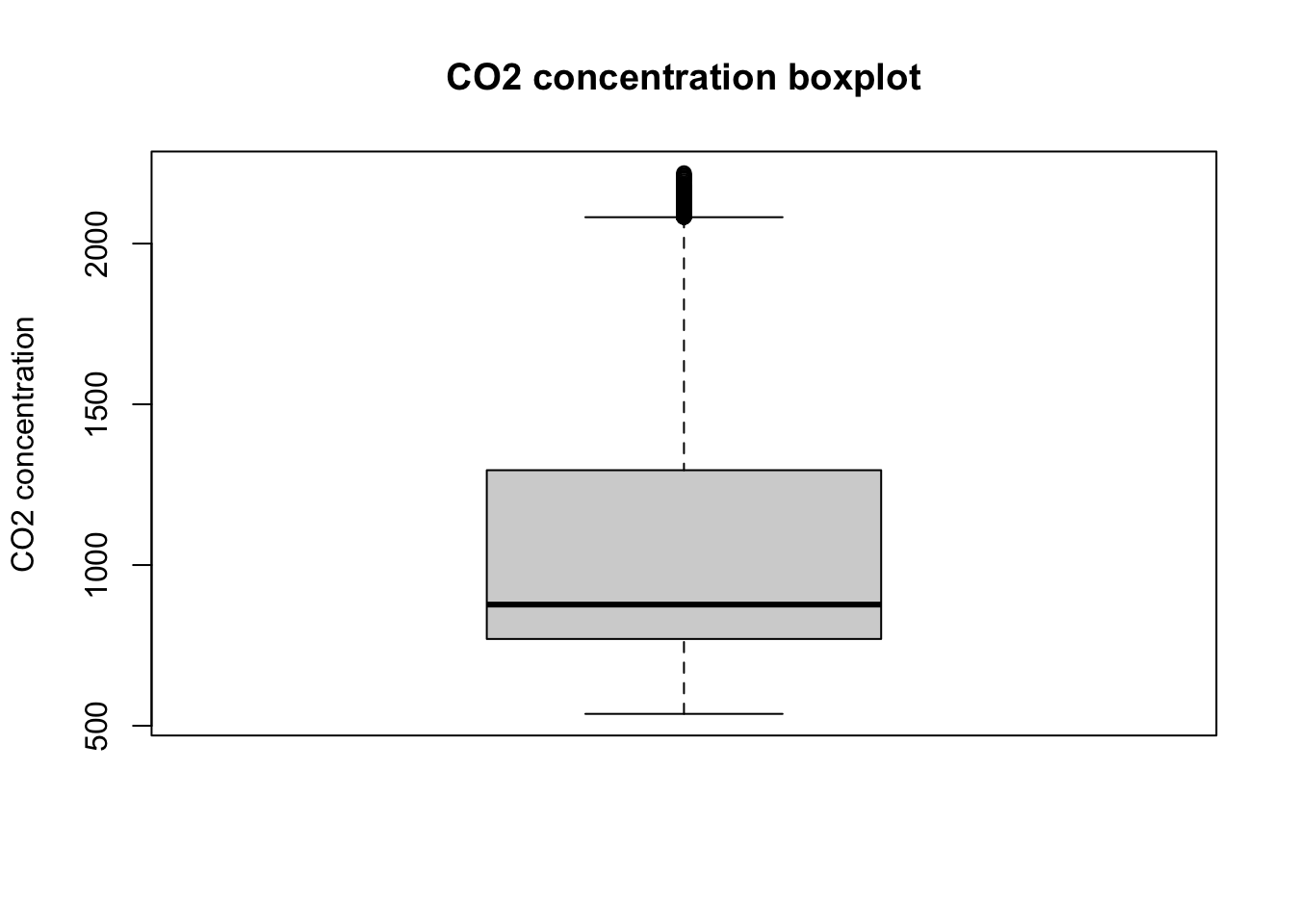
箱は四分位数を表し、箱の中の線は中央値を表します。 ひげは四分位範囲の1.5倍の範囲を表しその範囲を超える値は外れ値と言います。 点で示されている外れ値を取り除くには次のようにします。
co2 <- co2[co2$co2 >= bp$stats[1] & co2$co2 <= bp$stats[5], ]過去のAMeDASデータは、気象庁 > 各種データ・資料 > 過去の気象データ・ダウンロードから取得できます。 ファイル形式を参考にして読み解いていきます。 地点や変数等を選択します。 変数が多かったり、期間が長すぎたりすると、サイズの上限に達してしまいます。 その場合は、必要な変数や期間を絞ってください。 複数の地点を選択することもできますが、ここでは1地点を選んでください。 ここでは、一部に欠測とみなすデータが含まれている、四日市における降水量の合計(mm)、最高気温(℃)、日照時間(時間)について、2017年1年分の日別値を取得しました。
読み込んだデータはRを使って整理することができます。 例を見てみましょう。
列の名前は後でデータから拾ってつけるので、header=FALSEとします。 第1~3行目はダウンロードした時刻、空行、地点名などですので、skip=3で無視します。 R 4.2からはUTF-8が標準になりました。 AMeDASデータのファイル形式はShift JIS(cp932)なので、fileEncoding="cp932"を渡します。
raw <- read.csv("data.csv", header=FALSE, skip=3, fileEncoding="cp932")
head(raw) V1 V2 V3 V4 V5
1 年月日 降水量の合計(mm) 降水量の合計(mm) 降水量の合計(mm) 降水量の合計(mm)
2
3 現象なし情報 品質情報 均質番号
4 2017/1/1 0 1 8 1
5 2017/1/2 0 1 8 1
6 2017/1/3 0.0 0 8 1
V6 V7 V8 V9 V10
1 最高気温(℃) 最高気温(℃) 最高気温(℃) 日照時間(時間) 日照時間(時間)
2
3 品質情報 均質番号 現象なし情報
4 12.2 8 1 3.5 0
5 12.9 8 1 8.2 0
6 12.8 8 1 5.7 0
V11 V12
1 日照時間(時間) 日照時間(時間)
2
3 品質情報 均質番号
4 8 1
5 8 1
6 8 12行目のNAを消しておきます。 raw[3,] == "品質情報"は2行目の各要素が「品質情報」であればTRUE、それ以外はFALSEであるベクトルです。
raw[3, is.na(raw[3,])] <- ""
raw[3,] == "品質情報" V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12
3 FALSE FALSE FALSE TRUE FALSE FALSE TRUE FALSE FALSE FALSE TRUE FALSEこれを列の論理添字としてTRUEの列について7より大きいか調べ、論理ベクトルを作ります。 出力が長いので
head(raw[,raw[3,] == "品質情報"] > 7) V4 V7 V11
[1,] TRUE TRUE TRUE
[2,] FALSE FALSE FALSE
[3,] TRUE TRUE TRUE
[4,] TRUE TRUE TRUE
[5,] TRUE TRUE TRUE
[6,] TRUE TRUE TRUEapply()はRの強力な函数で、1番目の引数として渡す配列の次元(MARGIN、2番目の引数)に対して、3番目に渡す函数を適用します。 ここではallを渡します。
head(apply(raw[,raw[3,] == "品質情報"] > 7, 1, all))[1] TRUE FALSE TRUE TRUE TRUE TRUE均質番号と品質情報以外のデータの列を特定します。
!(raw[3, ] == "均質番号" | raw[3,] == "品質情報") V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12
3 TRUE TRUE TRUE FALSE FALSE TRUE FALSE FALSE TRUE TRUE FALSE FALSE品質情報が全て8(正常値)である行、データが含まれる列を残します。
filtered <- raw[apply(raw[,raw[3,] == "品質情報"] > 7, 1, all),
!(raw[3, ] == "均質番号" | raw[3,] == "品質情報")]
head(filtered) V1 V2 V3 V6 V9
1 年月日 降水量の合計(mm) 降水量の合計(mm) 最高気温(℃) 日照時間(時間)
3 現象なし情報
4 2017/1/1 0 1 12.2 3.5
5 2017/1/2 0 1 12.9 8.2
6 2017/1/3 0.0 0 12.8 5.7
7 2017/1/4 0.0 0 12.9 3.4
V10
1 日照時間(時間)
3 現象なし情報
4 0
5 0
6 0
7 01行目と2行目をつなげて列の名前として使います。
names(filtered) <- paste(filtered[1,], filtered[2,])
names(filtered)[1] "年月日 " "降水量の合計(mm) "
[3] "降水量の合計(mm) 現象なし情報" "最高気温(℃) "
[5] "日照時間(時間) " "日照時間(時間) 現象なし情報" Rで負の添え字は、その添え字を取り除くことを意味します。 Numpyのように後ろから数えるのではないことに注意しましょう。 1~2行目はデータではないので削除します。
filtered <- filtered[-(1:2),]
head(filtered) 年月日 降水量の合計(mm) 降水量の合計(mm) 現象なし情報 最高気温(℃)
4 2017/1/1 0 1 12.2
5 2017/1/2 0 1 12.9
6 2017/1/3 0.0 0 12.8
7 2017/1/4 0.0 0 12.9
8 2017/1/5 0 1 10.0
9 2017/1/6 0 1 9.5
日照時間(時間) 日照時間(時間) 現象なし情報
4 3.5 0
5 8.2 0
6 5.7 0
7 3.4 0
8 7.7 0
9 7.6 0大気の中には主要成分だけでなく、量は少ないが重要な働きをする微量成分があります。 二酸化炭素は人為起源の排出が原因で増加し続けています。 その様子をグラフにしてみましょう。 気象庁のWord Data Centre for Greenhouse Gases https://gaw.kishou.go.jp/publications/global_mean_mole_fractionsから年平均csvデータ(Global annual mean mole fractions)のCO2ファイルをダウンロードし、作業ディレクトに保存します。
df <- read.csv("co2_annual_20221026.csv")
plot(df$year, df$co2.global.mean.ppm.)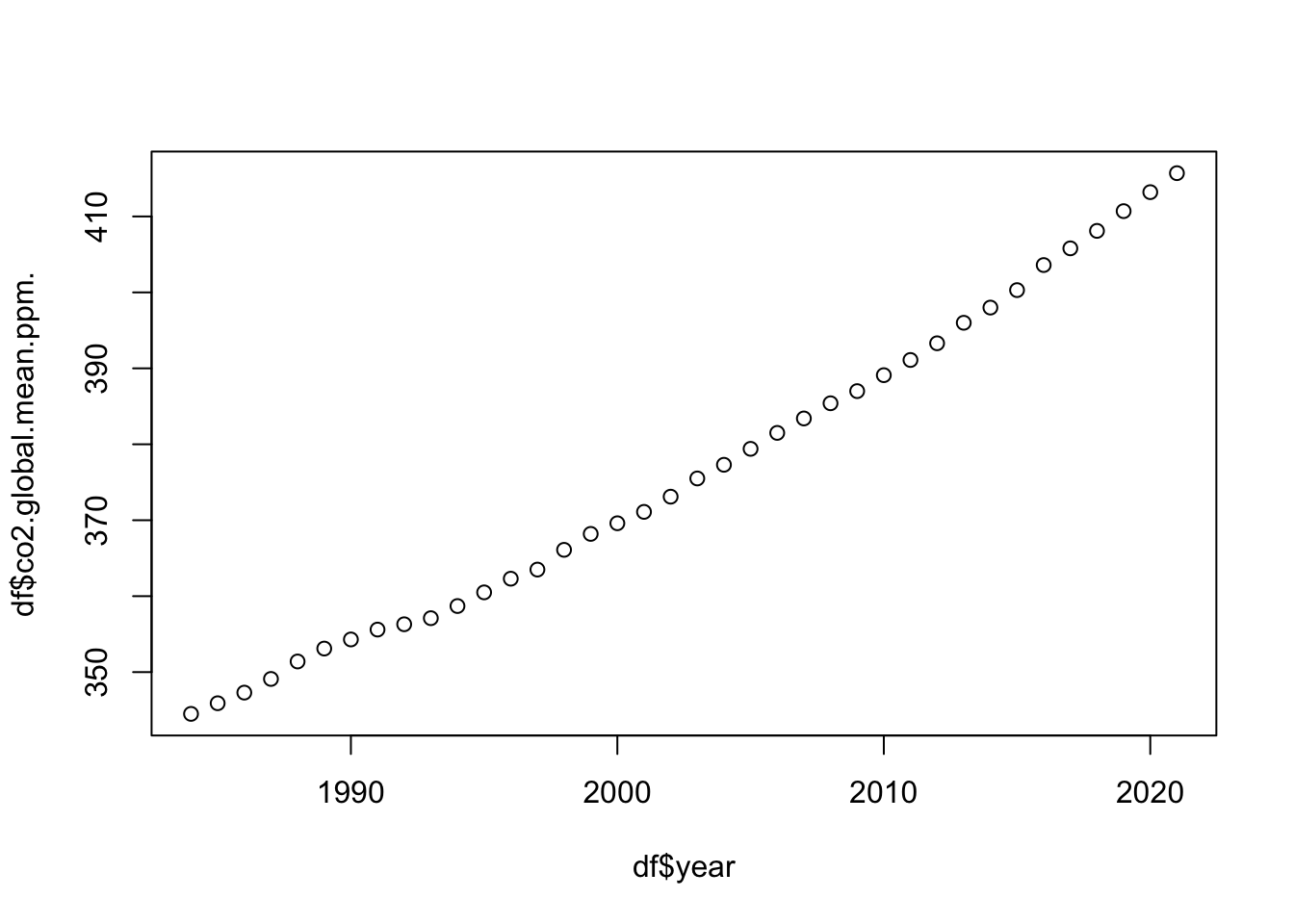
read.csv()はコンマ区切り(CSV, comma separated value)のデータを読む函数です。 ファイル名は文字列であることを表すために""で囲みます。 ファイル名の日付の部分は変わります。 グラフの種類を指定しないと、散布図になります。
dfには読んだ表の中身が入ります。dfはデータフレームと呼ばれるクラスのデータ構造です。データフレームは、値が行列に並んでいるだけでなく、行や列に名前がつけられます。
グラフにタイトルをつけ、軸のラベルを変更します。タイトルはmainで、軸ラベルはxlab、ylabで指定します。
plot(df$year, df$co2.global.mean.ppm.,
main="Global mean CO2 concentration",
xlab="year", ylab="CO2 ppm")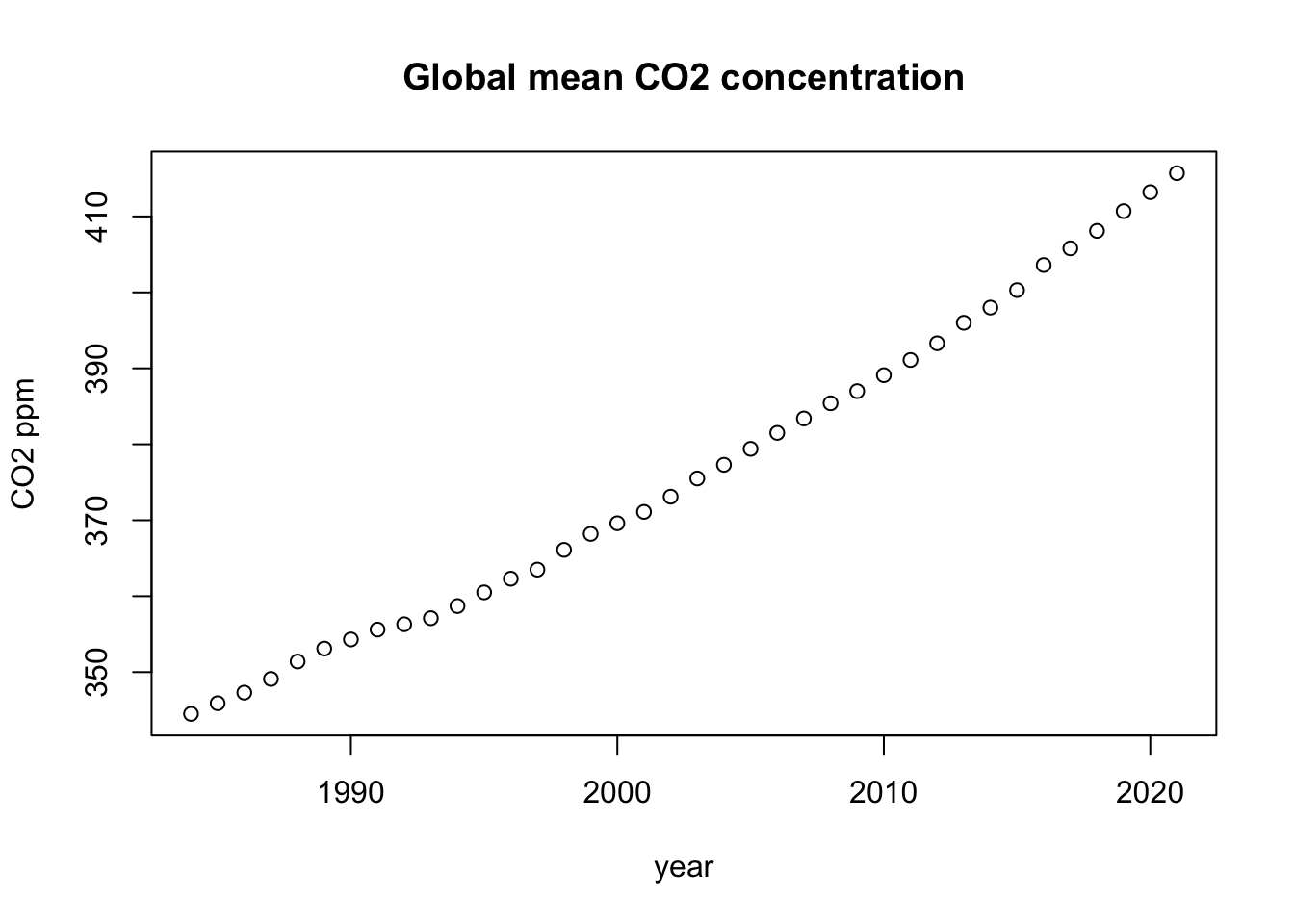
次に月平均データ(Global monthly mean mole fractions)を使って簡単な時系列解析をしてみます。
dfm <- read.csv("co2_monthly_20231115.csv")月毎の平均を求めてみます。
co2.annual.cycle <- aggregate(mole.fraction.ppm. ~ month, data = dfm, mean)
co2.annual.cycle month mole.fraction.ppm.
1 1 378.0431
2 2 378.4523
3 3 378.7062
4 4 378.7826
5 5 378.3562
6 6 377.1728
7 7 375.5664
8 8 374.6354
9 9 375.1223
10 10 376.7510
11 11 378.3100
12 12 379.3197プロットしてみましょう。
plot(co2.annual.cycle)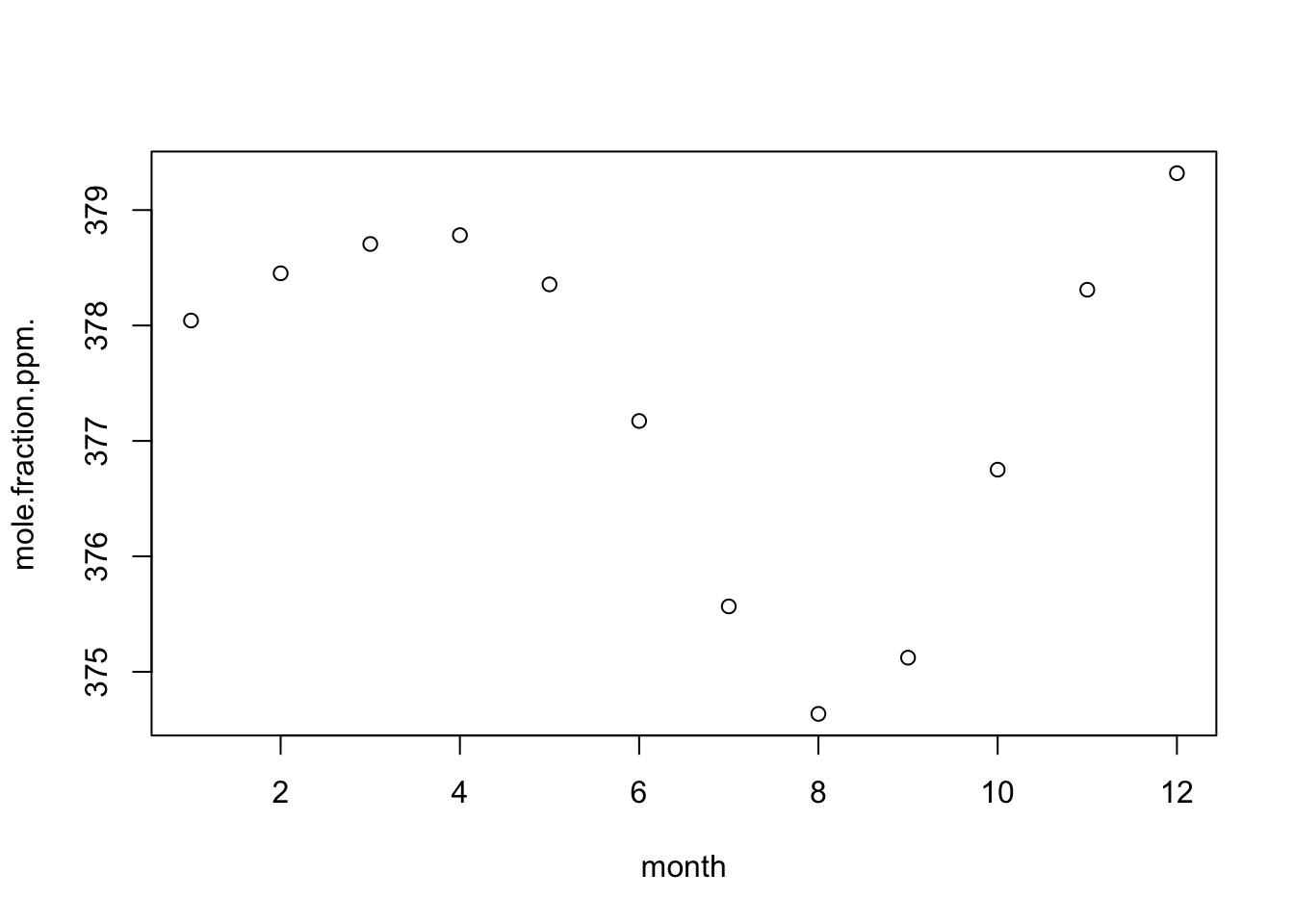
8月に一番少なく、1月に一番多くなっています。
co2.mon <- dfm$mole.fraction.ppm.
n <- nrow(dfm)
co2.mon <- ts(co2.mon, start=c(dfm$year[1], dfm$month[1]), frequency=12)
co2.mon.decomp<- decompose(co2.mon)
plot(co2.mon.decomp)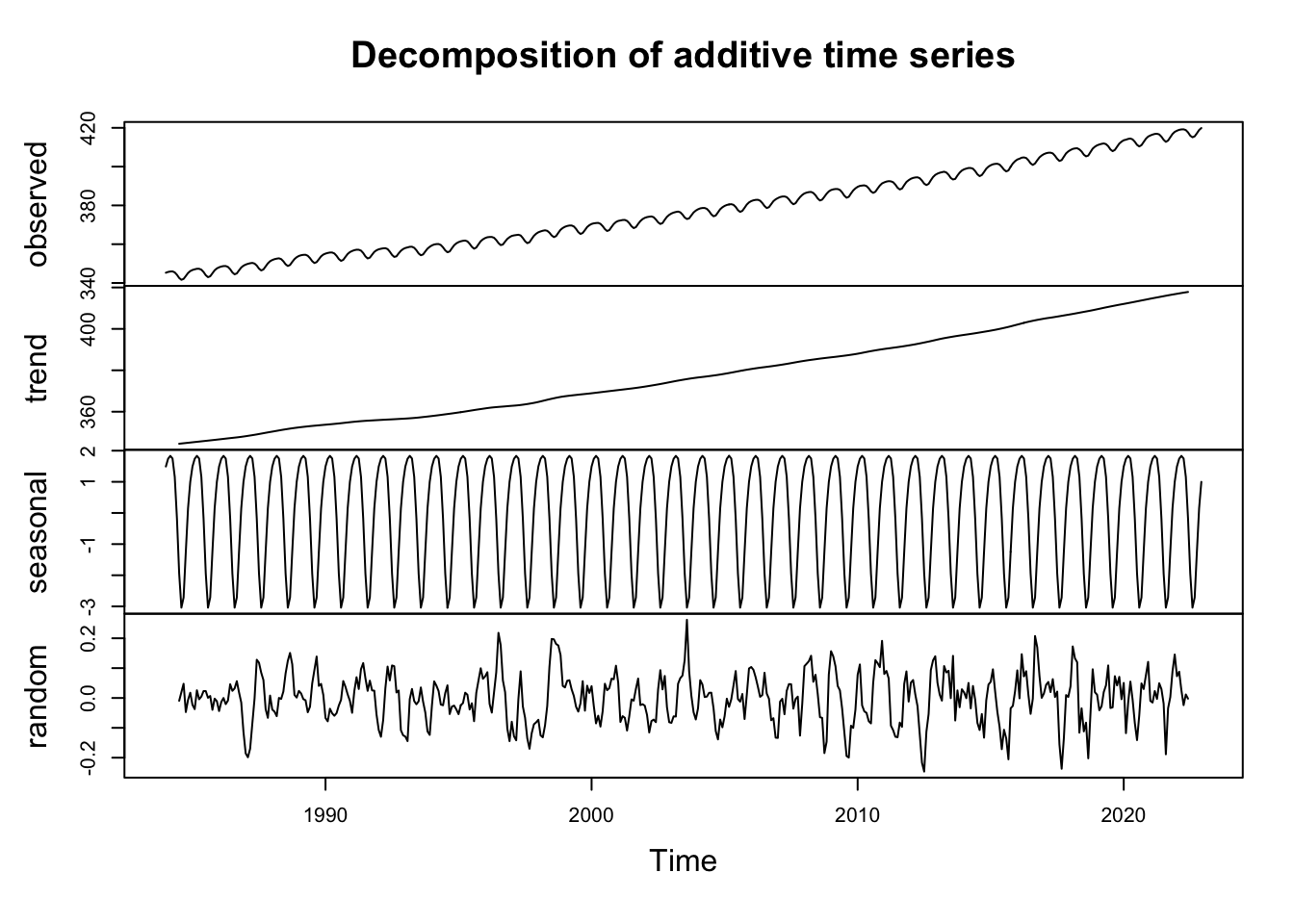
ts()は一つ目の引数のデータから時系列オブジェクトを作ります。 frequency=12は1年を単位として12回の頻度であることを示します。 decompose()はデータをトレンド、周期成分と残差に分解します。
残差をさらにスペクトル分解してみましょう。
spec.pgram(co2.mon.decomp$random, spans=c(7,7), na.action=na.omit)
abline(v=1)
abline(v=1/3)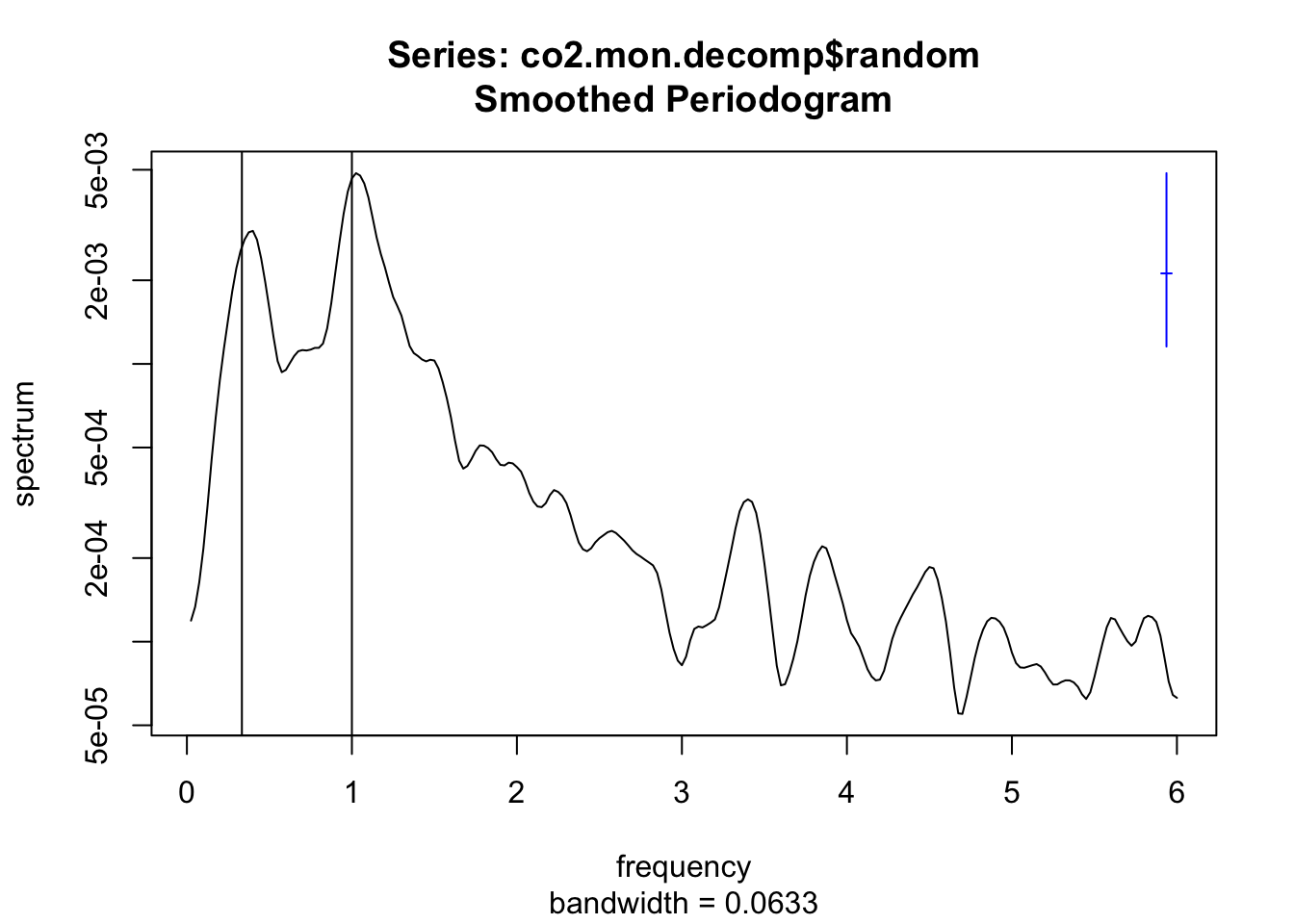
spec.pgramは高速フーリエ変換を利用して、ピリオドグラムを計算します。 spansで修正ダニエル法に基づいた平滑化を施すことができます。 右上の青い線は信頼区間を表します。 co2.mon.decomp$randomには最初と最後に数値でない値(not a number)をnaが入っていますので、na.actionで無視します。 ピークに近い、周期1年と3年に対応するところに縦線を入れてみました。 それぞれ年々変動とエルニーニョ現象のような数年周期に対応しているものと考えられます。
Rでは、このように対話的な簡単な操作により、簡単に解析やグラフの作成ができます。
自分で計測したCO2データをグラフにしてみましょう。
dfというテーブルがあり、dateという名前の列に2024/7/15、timeという列に12:00:00などのようにデータが入っているとします。これらをRの日時を表す型に変換して、列として追加するには次のようにします。
df$datetime <- as.POSIXct(paste(df$date, df$time))POSIXct型は1970年1月1日00:00:00からの秒数です。これを横軸に取ると良いでしょう。
気象庁は様々なデータをCSV形式で提供しています。 それらをRで読んでみましょう。 うまく読めるでしょうか。